![]()
N端乙酰化和肉豆蔻酰化是蛋白中最常见的翻译后修饰之一,它们参与了蛋白的相互作用和亚细胞定位。为了阐明这些翻译后修饰的重要性,需要比较分析修饰蛋白和未修饰蛋白。但是,在使用大肠杆菌等活细胞制备蛋白的常规方法中,受细胞中固有的修饰酶影响,难以控制和制备未修饰蛋白和修饰蛋白。
在本研究中,使用仅由参与蛋白合成的因子组成的PURE system(产品名称:PUREfrex®),探讨制备N端修饰目标蛋白的方法,并以完全可控的状态成功合成了乙酰化或肉豆蔻酰化的蛋白。
在使用常规PURE system的蛋白合成中,与大肠杆菌内的翻译反应相同,由甲酰化的蛋氨酸进行翻译。但是,如果初始蛋氨酸的氨基被甲酰化,则不会发生N端修饰。因此,使用不含甲酰基供体(10-Formyl-tetrahydrofolate10-CHO-THF)的PURE system进行蛋白合成。结果表明,尽管不使用10-CHO-THF后合成效率有所降低,但仍成功合成了目标蛋白。
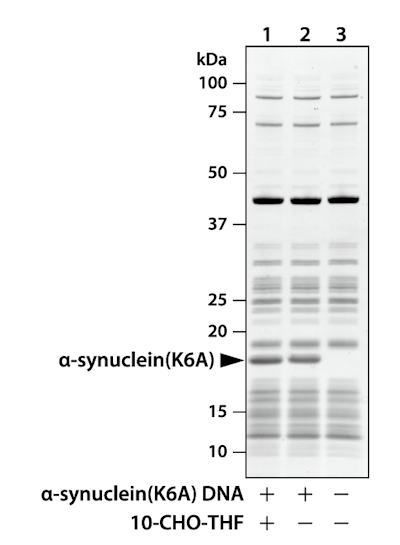
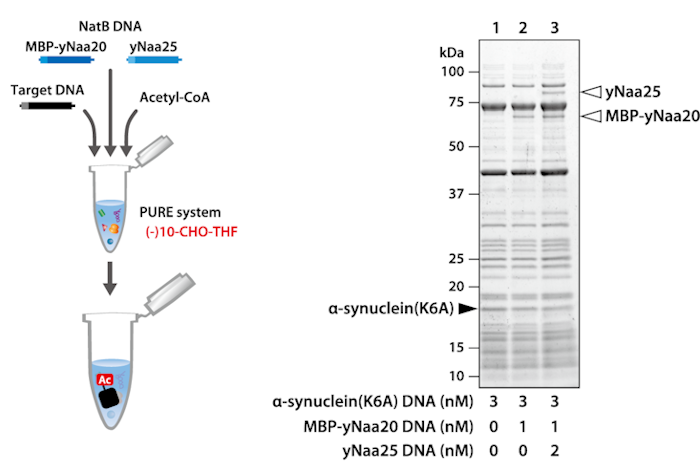
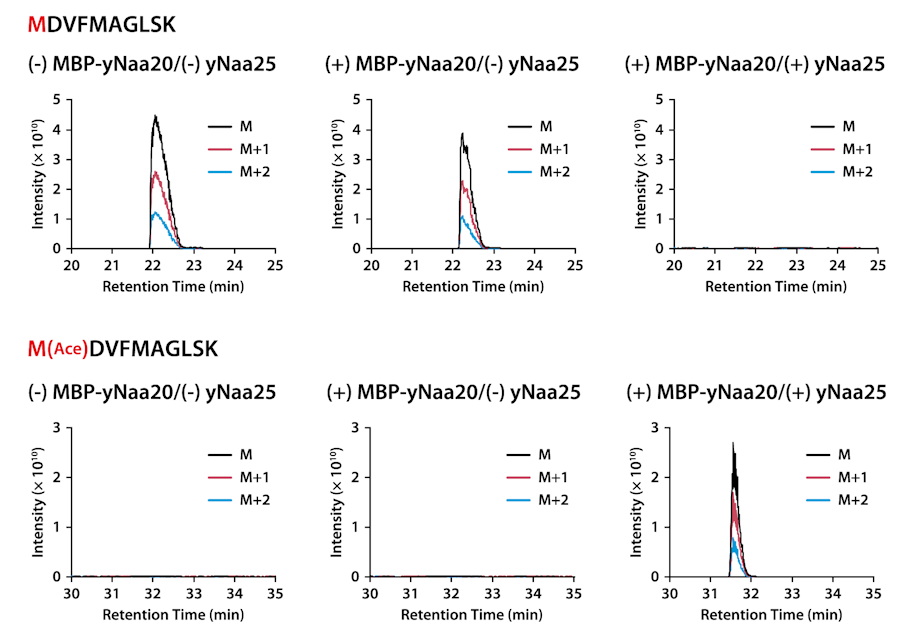
酵母NatB是一种由yNaa20和yNaa25组成的异源二聚体酶,它能将乙酰CoA的乙酰基转移至未甲酰化的初始蛋氨酸的氨基上。在不含10-CHO-THF的PURE system中加入目标蛋白、yNaa20及yNaa25的模板DNA和乙酰CoA进行反应。通过质谱分析合成的目标蛋白的N端肽,确认添加NatB DNA后被乙酰化。
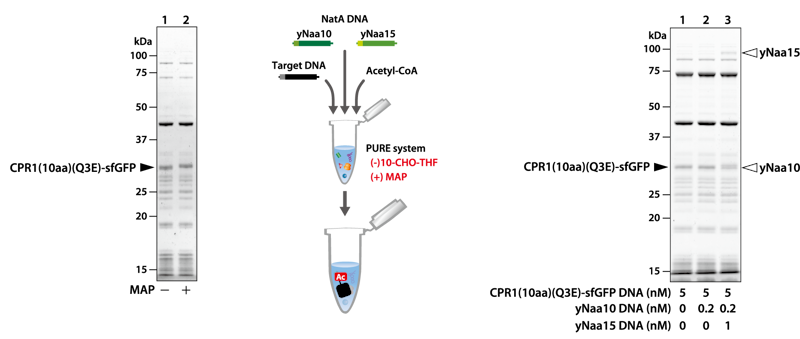
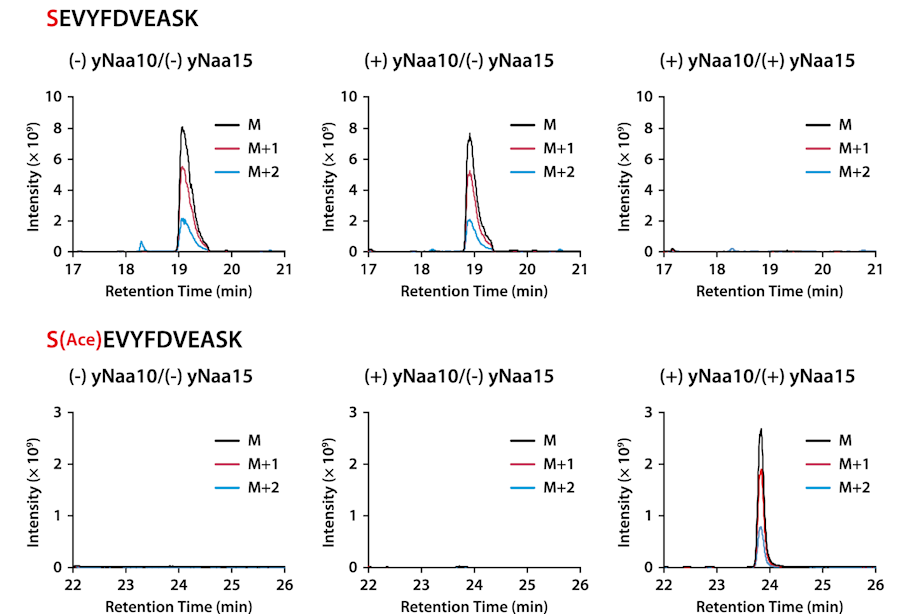
酵母NatA是一种由yNaa10和yNaa15组成的异源二聚体酶,它将乙酰CoA的乙酰基转移至methionine aminopeptidase(MAP)切割初始蛋氨酸时暴露的第二个氨基酸的氨基上。因此,使用NatA进行乙酰化时,必须通过MAP去除N端的初始蛋氨酸。由于MAP会切割未甲酰化的初始蛋氨酸,因在不含10-CHO-THF的PURE system中添加目标蛋白、yNaa10、yNaa15的模板DNA、纯化MAP和乙酰CoA进行反应。通过质谱分析合成目标蛋白的N端肽,确认添加NatA DNA后被乙酰化。
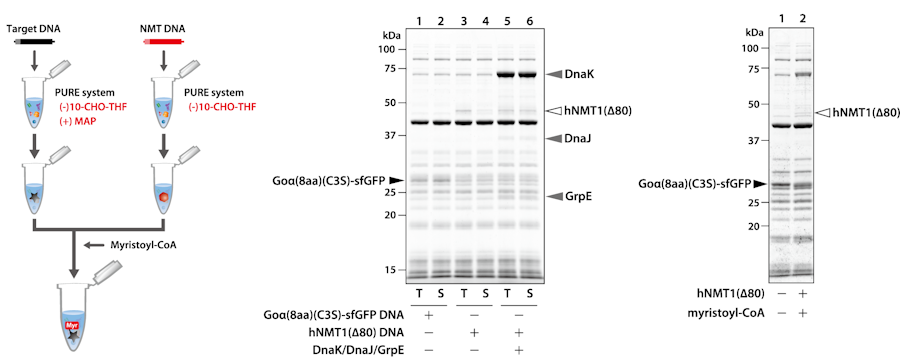
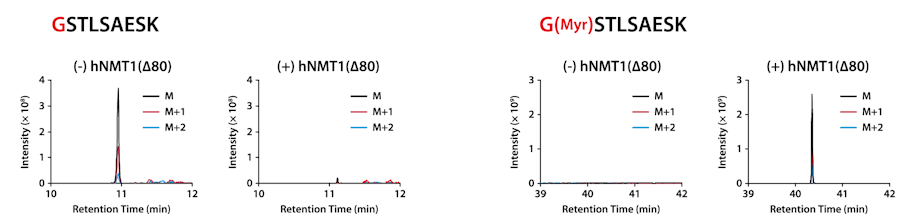
NMT是一种可将MAP切割初始蛋氨酸后暴露出的第二个甘氨酸的氨基进行肉豆蔻酰化的酶。由于底物肉豆蔻酰 CoA会抑制蛋白合成反应,因此需先在不含10-CHO-THF的PURE system中分别合成目标蛋白和NMT。然后,将分别合成的目标蛋白、NMT及肉豆蔻酰CoA混合,进行肉豆蔻酰化反应。通过质谱分析合成目标蛋白的N端肽,确认添加NMT后被肉豆蔻酰化。
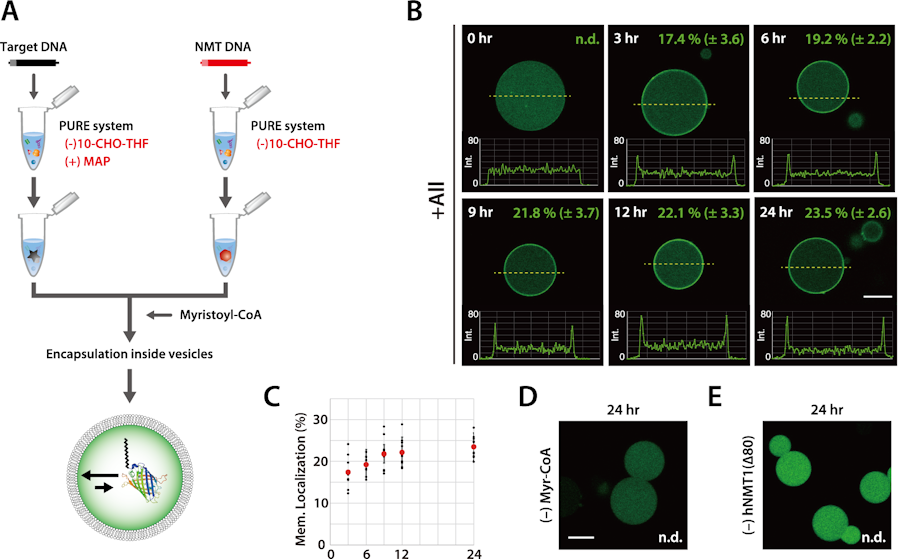
此外,在脂质体中进行上述的肉豆蔻酰化反应时,可观察到肉豆蔻酰化的蛋白在脂质体膜的定位状态。
● 实验步骤概述(A)
● 去除初始蛋氨酸的GFP通过hNMT1(Δ80)在脂质体内被肉豆蔻酰化,还可观察到其向脂质体膜迁移(B)
● 根据3、6、9、12和24 h的反应时间绘制GFP膜定位的比率(C)
● 未添加肉豆蔻酰-CoA(D)或hNMT1(Δ80)基因(E)时,无法观察到GFP的膜定位
该项研究结果表明,通过使用PURE system,可在完全可控下制备N端修饰蛋白,还可以确认翻译后修饰的合成蛋白在脂质体膜上的定位。此外,PURE system还能合成修饰酶,无需纯化即可用于修饰反应。该方法也适用于其他修饰酶,可以简便地进行各种翻译后修饰反应的分析和应用。
该项研究是GeneFrontier、东京工业大学和海洋研究开发机构(JAMSTEC)的共同研究。论文发表在ACS Synthetic Biology,可通过以下链接免费下载。
“Regulated N-Terminal Modification of Proteins Synthesized Using a Reconstituted Cell-Free Protein Synthesis System”
ACS Synthetic Biology (2023) 12, 1935-1942
相关产品:
| 免责声明 |
|
1. 本公司密切关注本网站发布的内容,但不保证发布内容的准确性、完整性、可靠性和最新性等。 2. 本公司不保证使用本网站期间不会出现故障或计算机病毒污染的风险。 3. 无论何种原因,使用本网站时给用户或第三方造成的任何不利或损害,本公司概不负责。此外,对于用户与其他用户或第三方之间因本网站发生的任何交易、通讯 3. 或纠纷,本公司概不负责。 4. 本网站可提供的所有产品和服务均不得用于人体或动物的临床诊断或治疗,仅可用于科研等非医疗目的。如任何用户将本网站提供的产品和服务用于临床诊断或治 4. 疗,以及其他特定的用途或行为,本公司概不保证其安全性和有效性,并且不负任何相关的法律责任。 |